Cosmo Bio would like to acknowledge and thank the Lexogen GmbH for providing “RNA Sequencing in Drug Discovery and Development” information presented here.
SLAMseqを用いたハイスループット kinetic RNAシーケンシング
SLAMseq 法 (RNA の代謝配列決定のためのチオール (SH) 結合のアルキル化) は、Ameres lab (Herzog et al., 2017) によって開発された方法で、生化学的抽出を必要とせずに、同じサンプルから新しく合成された (新生) RNA と既存の RNAを同時に同定して定量することができます。
SLAMseq とRNA-Seq を組み合わせることで、ハイスループットな動態 RNA シーケンス、またはハイスループットの時間分解 RNA シーケンスが可能になります。これは、細胞培養実験において、 RNA 動態に対して速効性のある薬剤または薬剤候補の効果を研究するのに理想的です。Kinetic RNA-Seq は、時間分解法で同化 (RNA 合成) または異化 (RNA 分解) プロセスを評価し、従来の方法よりもはるかに高い解像度で治療用化合物の有意な因果関係を明らかにすることができます。
SLAMseq は、なぜ創薬研究に最適なのか?
SLAMseq は、RNA 合成と RNA 分解を個別に測定し、薬物投与に応じた新しく生じた RNAの 発現や転写物の安定性を捕捉することができます。時間の次元が追加されると、一次または二次転写ターゲットが特定され、オフターゲットを検出することができます。さまざまな薬剤の効果を理解することは、起こりうる副作用の予測、薬物相互作用の検討、有効性の最適化に役立ちます。SLAMseqはがん研究に広く用いられており(Paris et al.,2019; Zhang et al.,2022)、低分子薬剤、rapid RNA療法薬、生物製剤などの速効型薬剤に適用することができます。
SLAMseq を使用して一次標的と二次標的を区別できる機能は、ターゲットの同定、潜在的なターゲットの検証、リードの最適化など、医薬品開発の様々なステップで有用です。SLAMseq を用いることで、生細胞内の転写プロセスをバイアスのない全体像で把握することができるため、あらゆる創薬研究に付加価値を与えることでしょう。
SLAMseqを用いたハイスループットkinetic RNA-Seqは、薬物の作用機序を明らかにし、疾患の存在や重症度を示す可能性のあるバイオマーカーの同定をサポートし、個別化医療や疾患の進行のモニタリングの原動力となりつつあります。SLAMseqは、バイオマーカーの同定をサポートすることで、新しい治療法の開発を加速し、疾患の治療成績の改善につながる可能性があります。
SLAMseqはどのように機能するのか?
SLAMseq法は、細胞に取り込まれやすいヌクレオチド類似体である4-チオウリジン(S4U)によるRNAの代謝標識と、カルボキシアミドメチル基を付加してS4U含有ヌクレオチドの4-チオール基を修飾するチオール反応性化合物ヨードアセトアミド(IAA)を用いたチオール(SH)結合アルキル化を組み合わせたものです。Lexogen の SLAMseq は、特許取得済みの S4U 標識技術と合理化されたワークフローを組み合わせた唯一の市販キットで、大規模な創薬研究を強化および加速します。
RNA標識は、各細胞タイプおよび培養方法に特異的です。新しい実験設定ごとに、SLAMseq では標識条件の最適化と S4U 取り込みの効率テストが必要です (探索段階)。RNA 動態解析では、RNA 合成 (同化) または RNA 分解 (異化) 速度を測定・モニタリングし、治療反応プロファイルを生成します。各サンプルは QuantSeq 3′ mRNA-Seq で前処理され、SLAMdunk 分析パイプラインを使用して配列決定およびデータ分析されます。SLAMdunk 分析パイプラインは、下流分析用に T>C を含むリードのリード数をレポートできるように特別に調整された修正アライメントアルゴリズムです (Herzog et al.、2017)。 (図1)。
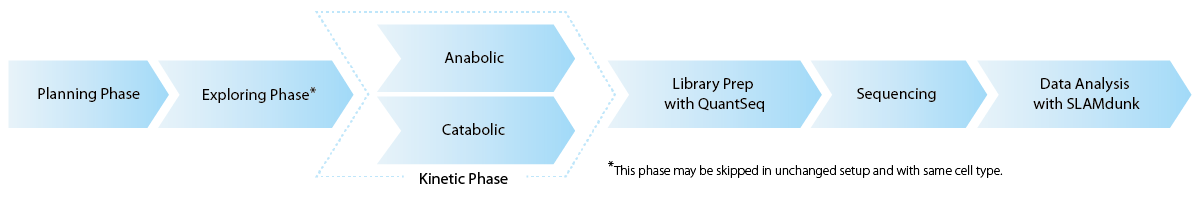
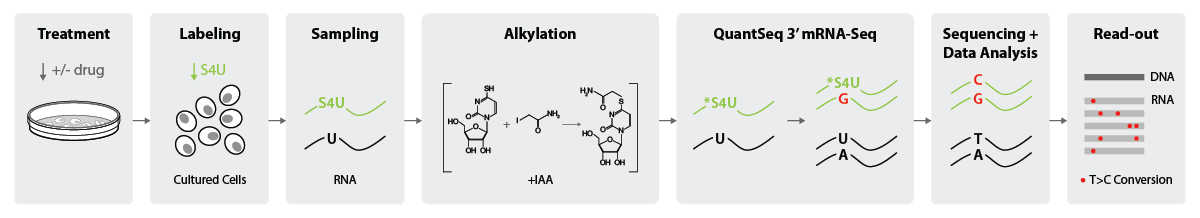
SLAMseq ラボ実験ワークフロー
SLAMseq は、培養細胞で使用するように設計されています (Herzog et al., 2017; Muhar et al., 2018)。RNA合成を評価するために、培養細胞はまず薬物またはコントロールで処理され、続いて培養培地に4-チオウリジン(S4U)が添加されます。S4Uは、新しく合成された転写産物に組み込まれ、それによってそれらを標識します。事前に選択した特定の時点で、細胞を採取し、RNAを単離し(サンプリングステップ)、ヨードアセトアミド(IAA)でアルキル化します。RNA-Seq ライブラリーは、QuantSeq 3′ mRNA-Seq を使用して生成され、逆転写酵素が還元された *S4U 修飾ヌクレオチドに遭遇する位置に A の代わりに G を組み込むため、逆転写中にヌクレオチド変換が行われます。シーケンシングにより、S4U 標識転写産物のチミンからシトシンへの (T>C) 変異が検出され、これにより新生 RNA レベルの変化のバイオインフォマティクス分析が可能になります (図 2)。
このワークフローは、RNAEXPERTiseで公開されているSLAMseqの動画をご参照ください。
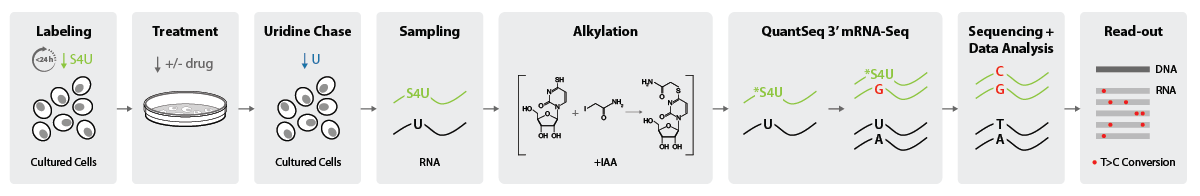
RNAのターンオーバーを評価する場合、実験の上流部分(サンプリングまで)は異なります。ここでは、パルスチェイスアプローチが使用され、細胞は最初に飽和に達するまで最大24時間S4Uで標識されます。次のステップは、細胞に薬剤等の処理を行い、インキュベーション後にウリジンを追加します。その結果、S4U信号は時間の経過とともに減少します(図3)。
まとめ
RNA-Seq は、トランスクリプトームの非常に動的な性質に対する理解に革命をもたらし、生物学、医学、臨床研究において実質的にかけがえのないツールとなっています。本稿で概説したように、治療に応じた差次的遺伝子発現に関する洞察の提供は、選択的スプライシング、アイソフォーム、遺伝子融合、低分子RNA、転写後修飾、scRNA-Seqによるシングルセルトランスクリプトミクスの調査、RNA動態の解明に至るまで、RNA-Seqとその多くのテクノロジーが新しい疾患生物学的に特定し、バイオマーカーをプロファイリングすることで、創薬・開発を大幅に補完する可能性を秘めています。RNA-Seqは、創薬と開発のさまざまな段階で使用され、次の目的で使用されます。
- 疾患の分子機構の理解
- 潜在的な薬剤ターゲット遺伝子の同定
- バイオマーカーの探索
- 薬物耐性と感受性に関与する遺伝子の同定
- 薬物毒性の評価
- 薬剤のリポジショニングの評価
- 薬理ゲノミクス
- テーラーメイド医療の開発
さらに深く掘り下げ、薬物の直接的および間接的な効果の本質的な詳細を真に理解するには、ハイスループット kinetic RNAシーケンシング用の定評あるSLAMseqテクノロジーを使用した時間分解RNAシーケンシングを用いることも有用です。
関連情報
商品の詳細:SLAMseq Metabolic RNA-Seq Kit(コスモ・バイオ)
がん研究における時間分解RNAプロファイリングに関するウェビナー
Time-resolved RNA profiling for cancer research and beyond
■参考文献
- Ergin, S., Kherad, N., and Alagoz, M.202249 (2022). RNA sequencing and its applications in cancer and rare diseases. Molecular biology reports 49, 2325-2333. DOI: 10.1007/s11033-021-06963-0.
- Herzog, V.A., Reichholf, B., and Neumann, T.; Rescheneder, Philipp; Bhat, Pooja; Burkard, Thomas R.; Wlotzka, Wiebke; Haeseler, Arndt von; Zuber, Johannes; Ameres, Stefan L.2017 (2017). Thiol-linked alkylation of RNA to assess expression dynamics. Nature Methods. DOI: 10.1038/nmeth.4435.
- Hughes, J.P., Rees, S., and Kalindjian, S.B.; Philpott, K. L.2011162 (2011). Principles of early drug discovery. British journal of pharmacology 162, 1239-1249. DOI: 10.1111/j.1476-5381.2010.01127.x.
- Khatoon, Z., Figler, B., and Zhang, H.; Cheng, Feng201475 (2014). Introduction to RNA-Seq and its applications to drug discovery and development. Drug development research 75, 324-330. DOI: 10.1002/ddr.21215.
- Mercer, T.R., Gerhardt, D.J., and Dinger, M.E.; Crawford, Joanna; Trapnell, Cole; Jeddeloh, Jeffrey A.; Mattick, John S.; Rinn, John L.201130 (2011). Targeted RNA sequencing reveals the deep complexity of the human transcriptome. Nature Biotechnology 30, 99-104. DOI: 10.1038/nbt.2024.
- Muhar, M., Ebert, A., and Neumann, T.; Umkehrer, Christian; Jude, Julian; Wieshofer, Corinna; Rescheneder, Philipp; Lipp, Jesse J.; Herzog, Veronika A.; Reichholf, Brian, et al.2018 (2018). SLAM-seq defines direct gene-regulatory functions of the BRD4-MYC axis. Science (New York, N.Y.). DOI: 10.1126/science.aao2793.
- Nguyen, N., Jennen, D., and Kleinjans, J.202227 (2022). Omics technologies to understand drug toxicity mechanisms. Drug Discovery Today 27, 103348. DOI: 10.1016/j.drudis.2022.103348.
- Paris, J., Morgan, M., and Campos, J.; Spencer, Gary J.; Shmakova, Alena; Ivanova, Ivayla; Mapperley, Christopher; Lawson, Hannah; Wotherspoon, David A.; Sepulveda, Catarina, et al.2019 (2019). Targeting the RNA m6A Reader YTHDF2 Selectively Compromises Cancer Stem Cells in Acute Myeloid Leukemia. Cell Stem Cell. DOI: 10.1016/j.stem.2019.03.021.
- Saini, J.S., Corneo, B., and Miller, J.D.; Kiehl, Thomas R.; Wang, Qingjie; Boles, Nathan C.; Blenkinsop, Timothy A.; Stern, Jeffrey H.; Temple, Sally201720 (2017). Nicotinamide Ameliorates Disease Phenotypes in a Human iPSC Model of Age-Related Macular Degeneration. Cell Stem Cell 20, 635-647.e7. DOI: 10.1016/j.stem.2016.12.015.
- Shaheen, S., Fawaz, F., and Shah, S.; Büsselberg, Dietrich201819 (2018). Differential Expression and Pathway Analysis in Drug-Resistant Triple-Negative Breast Cancer Cell Lines Using RNASeq Analysis. International journal of molecular sciences 19. DOI: 10.3390/ijms19061810.
- Singh, N., Vayer, P., and Tanwar, S.; Poyet, Jean-Luc; Tsaioun, Katya; Villoutreix, Bruno O.20233 (2023). Drug discovery and development: introduction to the general public and patient groups. Front. Drug Discov. 3. DOI: 10.3389/fddsv.2023.1201419.
- Xu, J., Gong, B., and Wu, L.; Thakkar, Shraddha; Hong, Huixiao; Tong, Weida20168 (2016). Comprehensive Assessments of RNA-seq by the SEQC Consortium: FDA-Led Efforts Advance Precision Medicine. Pharmaceutics 8. DOI: 10.3390/pharmaceutics8010008.
- Yang, X., Kui, L., and Tang, M.; Li, Dawei; Wei, Kunhua; Chen, Wei; Miao, Jianhua; Dong, Yang202011 (2020). High-Throughput Transcriptome Profiling in Drug and Biomarker Discovery. Frontiers in Genetics 11, 19. DOI: 10.3389/fgene.2020.00019.
- Zhang, J., Wang, Y., and Zhen, P.; Luo, Xia; Zhang, Chao; Zhou, Lin; Lu, Yanxin; Yang, Yang; Zhang, Wei; Wan, Jun20138 (2013). Genome-wide analysis of miRNA signature differentially expressed in doxorubicin-resistant and parental human hepatocellular carcinoma cell lines. PLoS One 8, e54111. DOI: 10.1371/journal.pone.0054111.
- Zhang, Y., Remillard, D., and Onubogu, U.; Karakyriakou, Barbara; Asiaban, Joshua N.; Ramos, Anissa R.; Bowland, Kirsten; Bishop, Timothy R.; Ott, Christopher J.; Janiszewska, Michalina, et al.2022 (2022). Collateral lethality between HDAC1 and HDAC2 exploits cancer-specific NuRD complex vulnerabilities.
掲載元:LEXOGEN社ブログ
”RNA Sequencing in Drug Discovery and Development”


