2023年8月5日(土)、摂南大学農学部で開催された「遺伝子で調べよう!野菜のルーツ」という公開講座には、小学生から社会人まで幅広い年齢層の25名が参加し、野菜の遺伝子解析を通じて、野菜の類縁関係や進化について学びました。
前編では、野菜からDNAを抽出し、PCR反応でDNAを増幅し、電気泳動でその断片の大きさを確認しました。さらに、シークエンスによって各野菜の正確なDNA塩基配列を知るところまでご紹介しました。
後編では、得られた野菜の塩基配列を基に、相同性検索(DNAやタンパク質の配列をデータベースと照合し、類似する遺伝子や生物を探すこと)や類似度検索(配列間の類似度を計算し、進化的な近さを数値で示すこと)を行い、野菜同士の遺伝的なつながりを解明していきます。また、解析結果をもとに系統樹を作成し、野菜がどのような進化をとげているのかを明らかにしていきます。

▼摂南大学公開講座 2023 前編 – 遺伝子で調べよう!野菜のルーツ – はこちら
実験
実験3:シークエンス結果から植物の名前を検索する
日本のDDBJ(DNA Data Bank of Japan:日本の国立遺伝学研究所が運営する遺伝情報のデータベース)、欧州のEMBL(European Molecular Biology Laboratory:ヨーロッパ分子生物学研究所のこと)、米国のGenBank(米国の国立生物工学情報センターが運営する遺伝情報のデータベース)といった遺伝情報のデータベースには、さまざまな生物の遺伝情報が収集されています。今回の実験では、解析した野菜サンプルのDNA塩基配列をこういったデータベースと照合し、BLAST検索によって種名を特定します。
このような検索は相同性検索と呼ばれ、DNA配列の類似性を基に近縁種を探します。また、類似度検索では、配列同士の類似性を数値化し、進化的な近さを評価します。これにより、得られた情報をもとに、植物の進化的関係を示す系統樹を作成することができます。

こうした遺伝子やタンパク質のデータをコンピュータで解析する手法をバイオインフォマティクス解析といい、生物の類縁関係や進化を解明するために重要な役割を果たします。
3-1. シークエンス情報のフォルダを開いて波形を表示させる
パソコンを起動し、ENTERキーを押してログインする。デスクトップの「野菜DNA波形」フォルダを開き、サンプルファイルを1つ選んでダブルクリックする。
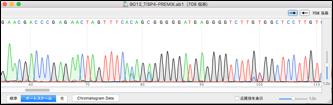

DNAの塩基配列は、A(アデニン)、T(チミン)、G(グアニン)、C(シトシン)の4種類で表されます。シークエンス機械が生成した4色のグラフは、それぞれの塩基を示しており、グラフの上に表示されているアルファベットの並びがDNAの実際の配列です。
3-2. 野菜のDNA塩基配列を探す
① CLC Sequence Viewer(遺伝子解析やバイオインフォマティクスのために設計された無料のソフトウェア)を起動する。
②「野菜DNA」を選択する。
③ リストに表示された70種類の植物DNAから、自分が選んだ野菜のDNA塩基配列を探す。
3-3. 相同性検索をおこなう
① NCBIのBLAST(相同性検索)ページを開き、「Nucleotide BLAST」をクリックして下図の一番右側の画面を表示させる。
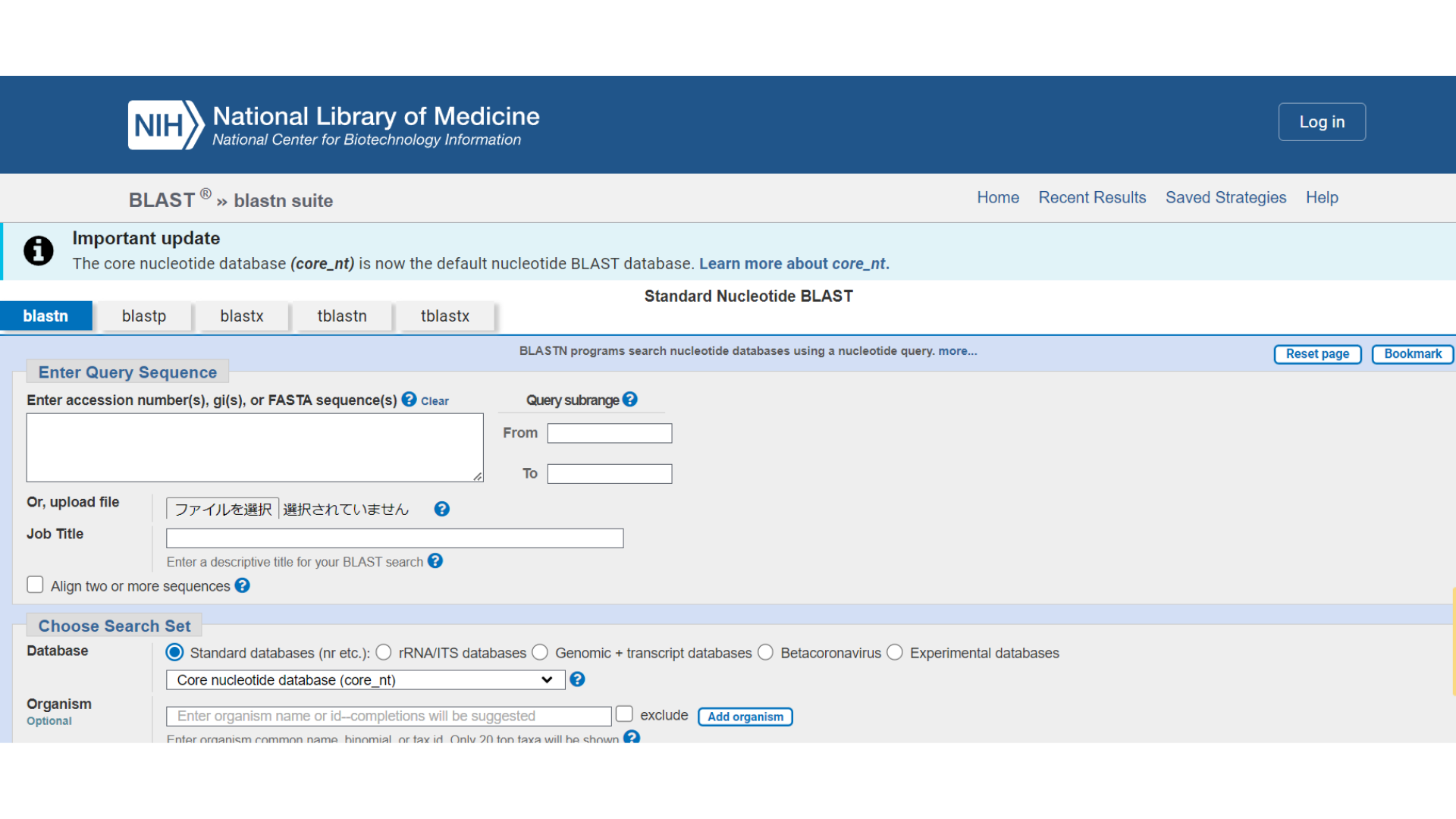
② CLC Sequence Viewerで選んだ野菜のDNA塩基配列をコピーし、Nucleotide BLASTの入力欄にペーストする。その後、ページ下部までスクロールして「BLAST」をクリックする。約2〜5分待つと、類似した塩基配列を持つ生物のリストが表示される。
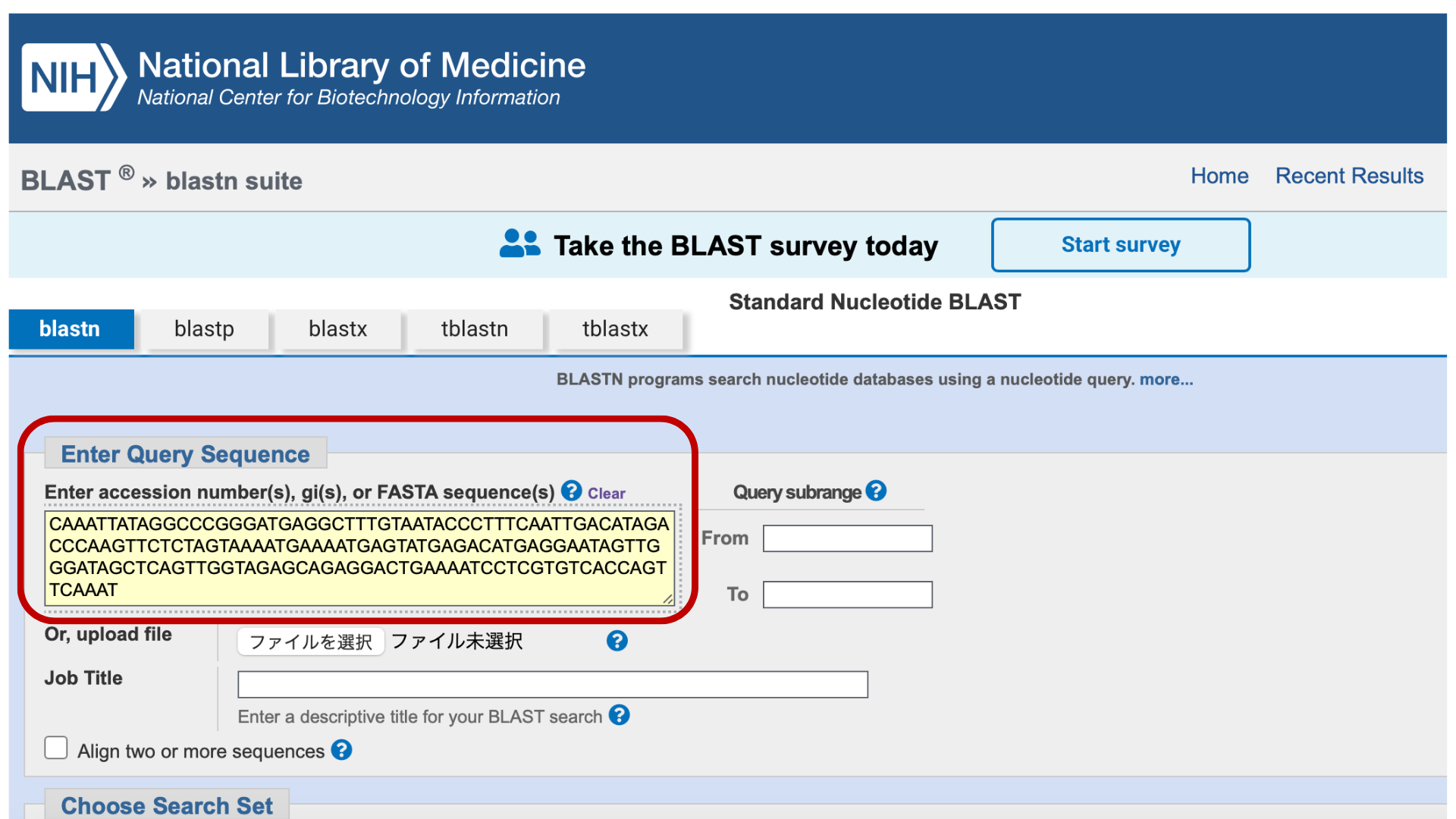
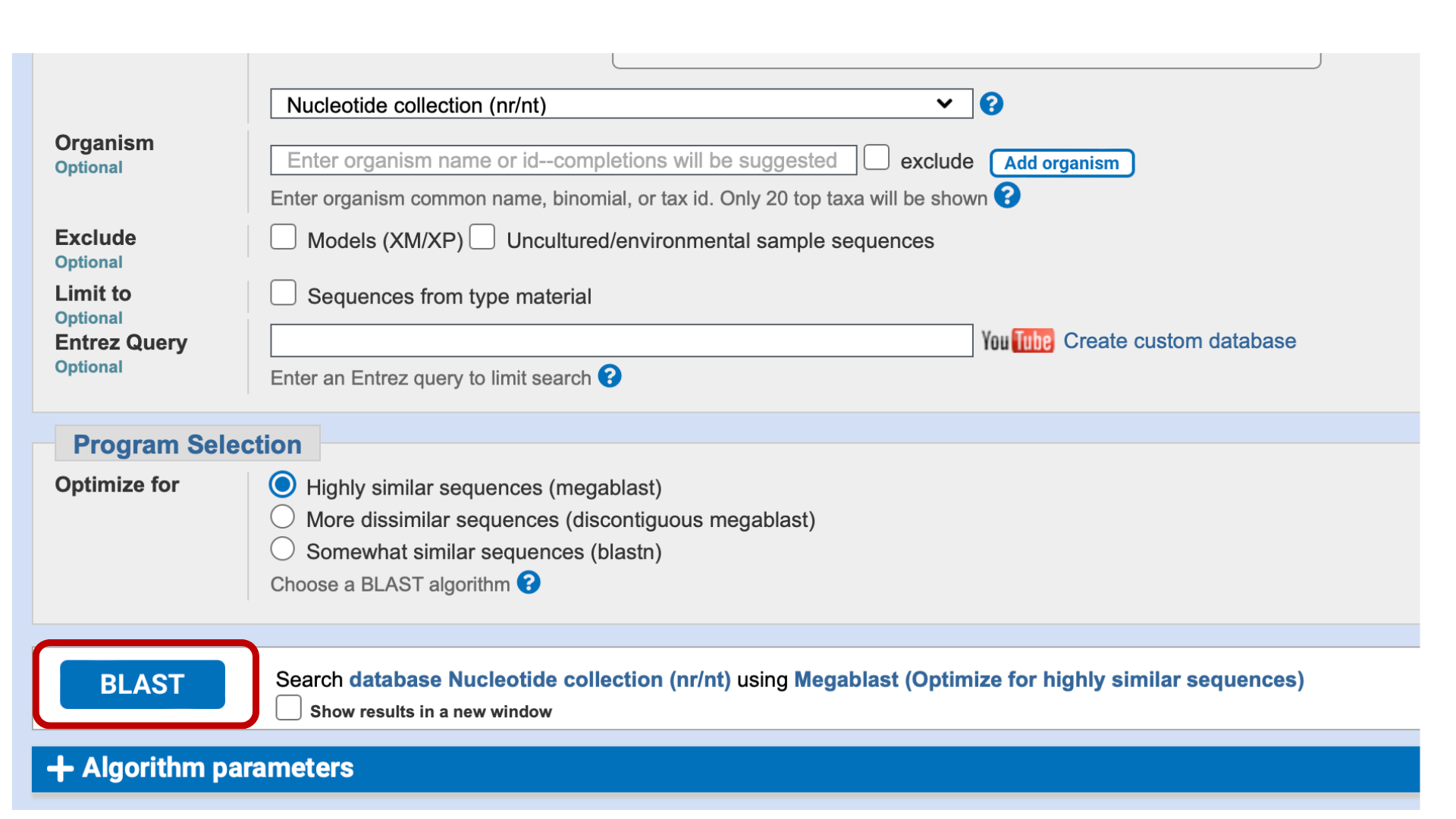

良く似た塩基配列を持つ生物ほど、リストの上位に表示されます。
③リストの一番上に表示された生物名をコピーして、Googleで検索し、一般名を調べる。自分が選んだ野菜名が出てくれば、相同性検索は完了。
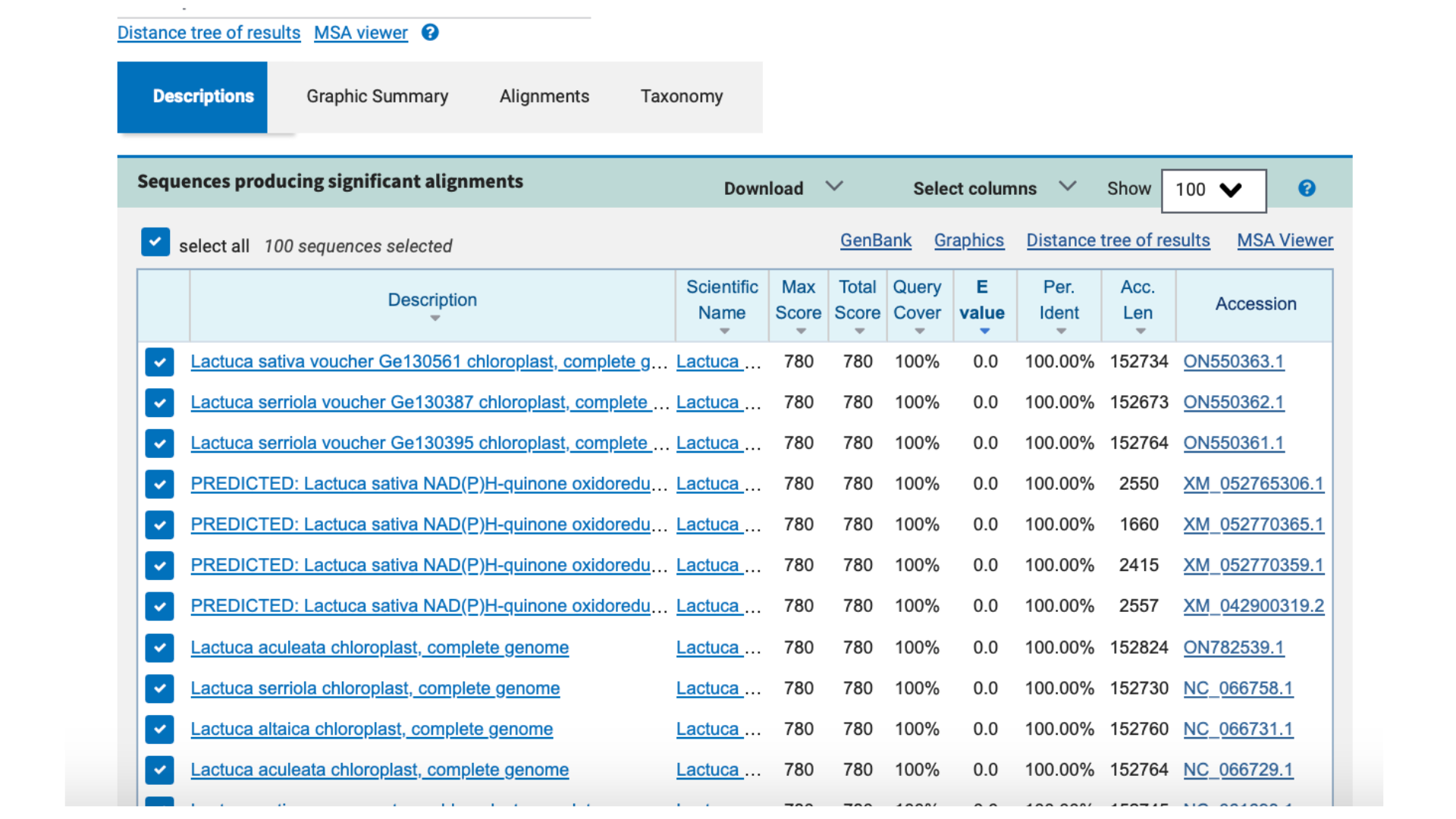

このように、遺伝的に親戚関係にある生物は非常に似た塩基配列を持っていることがわかります。こうやって遺伝的な類縁関係を明らかにしていきます。

NCBI(National Center for Biotechnology Information、米国国立生物工学情報センター)は、遺伝情報や生物医学データを収集・提供する米国の機関だよ。世界中の研究者がアクセスできるDNAデータベース(GenBankなど)を運営していて、BLASTなどの相同性検索ツールを提供してくれているよ。
実験4:系統樹を作成する
進化段階の異なる10種類の植物を基準として使用し、各自が選んだ野菜のDNA配列がこれらの植物とどのような関係にあるかを明らかにします。今回、基準とする植物として選んだのは以下です。

この植物たちを基に、野菜の進化の道筋を探る系統樹を作成します。
4-1: 基準とする植物のDNA配列を開き、整列させる
① スタートボタンからCLC Sequence Viewerを起動する。
② 基準となる植物のフォルダをクリックして開く。
③ 次に、フォルダ内から自分が選んだ野菜のファイルを開く。
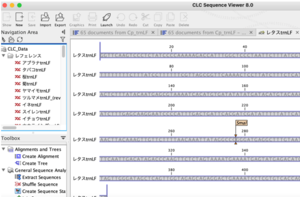
④ 左下のToolboxで「Alignments and Trees(整列図や樹形図)」フォルダを選び、「Create Alignments(整列図を作成)」を選択する。基準植物10種と自分が選んだ野菜のファイルを選び、右矢印をクリックして右側のウィンドウに移動させたら、「Next」をクリックする。

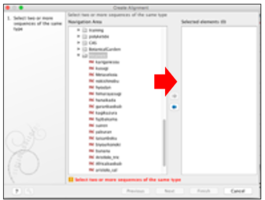
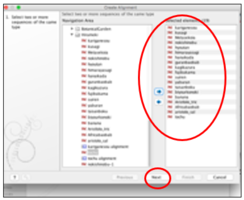
⑤ パラメータ設定は変更せず、そのまま「Next」をクリックする。次の画面で「Open」にチェックを入れ、「Finish」をクリックすると、アライメント(配列の整列)が完成する。
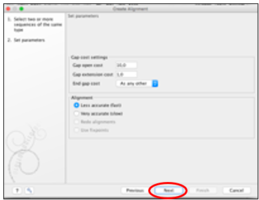
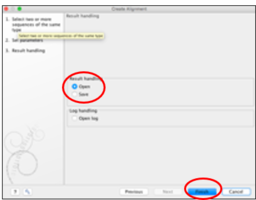
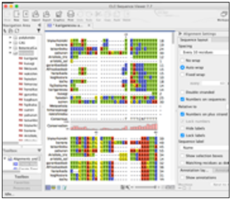
⑥ 完成したアラインメントに名前をつけて保存する(例: “reference” など)。
4-2: アラインメントから系統樹を作成する
① CLC Sequence Viewerの左下のToolboxからAlignments and Trees(整列図や樹形図)フォルダを選択し、「Create Tree」をクリックする。
② 4-1で作成したアライメントを右側のウィンドウに移動し、「Next」をクリックする。
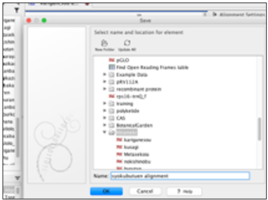

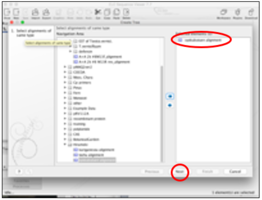
③パラメータはそのままで「Next」をクリックする。次の画面でOpenにチェックを入れ、「Finish」をクリックする。
④ 系統樹が完成する。

先生
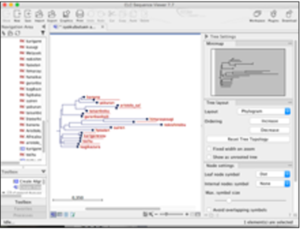
右の図のように、系統樹は枝の色やパラメータを変更して、好みの図にカスタマイズすることもできますよ。

先生
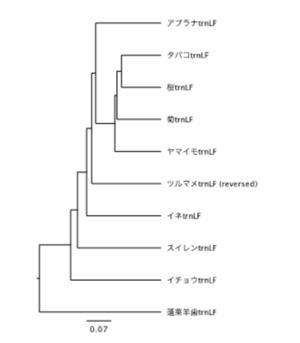
右の図に示されているのは、基準となる植物たちの系統樹です。皆さんが解析した植物も、この系統樹のどこかに近い位置にあるはずです。それが、皆さんの野菜に最も近い親戚にあたる植物なんですよ。



キャベツやハクサイ、レタス、タマネギといった結球野菜や、サンチュ、パセリ、ホウレンソウ、ミズナ、チンゲンサイ、ケール、ネギといった葉物野菜、そしてダイコン、ニンジン、ジャガイモ、サツマイモ、サトウダイコン(テンサイ)、サトイモ、ヤマイモ、ゴボウなどの根菜も加えて、系統樹を作ってみましょう!
どの植物がどの基準植物や他の野菜に近いか見つけられるでしょうか?見た目が似ている野菜でも、系統的には遠いものもあれば、意外な植物が近縁であったりします。
形が似ていても、実は異なる種類の植物があることに気づくかもしれません。植物学の本やインターネットで調べていくと、野菜への理解が深まり、もっと好きになるはずです。
アンケート結果
受講後のアンケートでは、プログラム内容がわかりやすく、とても興味深かったという結果が得られました。また、科学への興味や関心も高まったようです。



以下のような感想も寄せられました。
- 普段使えない機器を使って体験させていただき勉強になりました。最後は難しい所もありましたがいい経験ができ、また興味が膨らみました。またなにかあれば参加させて頂きたいと思います。
- 大学の研究室の装置や器具を使っての実験はなかなかできることではないので、小学生でも理解できるか?と思いながらも参加させていただいた。最初はピペット操作すらも難しそうだったが、実験が終わってから、行ってよかったと話していた。
- 遺伝子が近そうでも遠かったり、意外な発見ができて面白かったです。自分の手を動かして実験できたことは貴重な体験でした。中学生でも、わかりやすく興味深い内容でした。
- 講座の内容に興味があり、参加させていただきました。普段使うことのない実験器具や装置に触れて、貴重な体験になりました。先生のわかりやすい説明とヘルプの学生さんたちの丁寧な対応がすばらしく、有意義な時間をありがとうございました。また、機会がありましたら参加したいのでよろしくお願いします。
公開講座を終えて
今回の公開講座では、野菜のDNA抽出から始まり、PCR反応や電気泳動、そしてバイオインフォマティクス解析まで、多彩な実験を通じて野菜の遺伝的な類縁関係や進化の道筋を解き明かしました。
まず、オリエンテーションとして、実際の野菜を手に取って形や色で分類を試みるアイスブレイクを行い、予想が解答と異なる場面では驚きや発見の声が上がり、参加者の科学的好奇心を刺激する良いスタートになりました。
実験に入ると、マイクロピペットの操作に戸惑いながらも、子どもから大人まで真剣に取り組む姿が印象的でした。液体窒素を使った凍結や粉砕、ボルテックスミキサーや遠心分離機の操作を楽しむ様子からも、科学への興味がさらに高まっているのを感じました。透明なDNA溶液を目にした際には、「本当に取れているの?」と素直な疑問があがり、好奇心を持ちながら実験に向き合っている様子が伝わりました。
増幅したDNAの電気泳動や、青色光で確認する作業にも挑戦し、野菜ごとに異なるDNAバンドの大きさを学びました。結果をプリントアウトしてお土産として持ち帰ってもらい、ほとんどの方が実験に成功した達成感を感じているようでした。
最後に、バイオインフォマティクス解析により、各野菜のDNA配列を基に相同性検索を行って最も近縁の生物種を同定し、系統樹を作成しました。予想外の親戚関係に驚く声も上がり、日常で目にする野菜に新たな視点と興味が生まれたことでしょう。
今回の講座を支えてくださった先生方やティーチングアシスタントの皆さん、そして積極的に参加してくださった受講生の皆さんに心からの感謝とお疲れ様をお伝えします。この貴重な体験が、皆さんにとって科学への興味や学びのきっかけとなりますように。本当にお疲れ様でした!
【イラスト・画像の引用元について】
本記事内で使用されたイラスト・画像は、摂南大学様のご厚意により、同大学からご共有いただいた資料の一部を引用させていただいております。

