2023年8月5日(土)、摂南大学農学部8号館にて公開講座が開催されました。小学生から社会人まで、幅広い層の25名が参加しました。
私たちが普段食べている野菜は、形や色が様々です。キャベツとレタス、大根とニンジンは形が似ている一方、紫キャベツとナスは色が似ています。しかし、見た目だけでは野菜の類縁関係を判断するのは難しいものです。
この講座では、こうした身近な野菜を使い、DNA分析とコンピュータ、データベースによる遺伝情報解析をおこなうことで、野菜の類縁関係を調べました。また、野生の植物としての野菜のルーツも解析し、その進化の過程にふれました。

講師紹介
この講座の講師陣は、摂南大学農学部応用生物科学科の椎名隆教授、加藤裕介准教授、石崎陽子助手です。皆さん、植物分子生物学のプロフェッショナルとして活躍されています。
また、摂南大学の4回生3名、3回生3名のティーチングアシスタント(TA)の方がサポートに加わり、受講者の実験を支えてくれました。

普段食べている野菜に、どのような遺伝的なつながりがあるのかを、科学的な手法を学びながら知ってもらえたら嬉しいです。そして、DNAを通じて、野菜がどのように進化してきたのかをひも解いていきましょう。遺伝学的な視点から身近な野菜をみる楽しさ、そして知る喜びを一緒に味わいましょう!
実験
ウォーミングアップ:野菜を仲間に分けてみよう
実験の前に野菜の形やルーツに関する解説が行われました。その後、参加者は1人ずつ野菜を7つのグループに分け、簡単な自己紹介とともに、なぜそのように分類したのかをチームで話しました。また、DNA塩基配列を手がかりにすると、どのようにグループ分けが変わるのかを予想しました。

科学は、仮説を立て、それを確かめていく中で多くの発見が生まれます。「この野菜はこの仲間かもしれない」という予想が、実験結果との違いを際立たせ、新しい発見につながります。予想を立てながら実験に取り組みましょう。
実験1:野菜からのDNA抽出とPCR反応
実験1では、野菜からDNAを抽出し、それをPCR反応で増幅します。細胞は糖類、脂質、たんぱく質、DNA、RNAなどで構成されていますが、DNAを正確に分析するためには、DNA以外の成分を取り除き、純粋なDNAを抽出する必要があります。
はじめに、-196℃の液体窒素で野菜を凍らせて粉砕し、DNA抽出バッファーでDNAを含む成分を水に溶かします。次に、タンパク質変性剤を使ってタンパク質や糖類を除去し、最後はカラム(DNAを吸着する樹脂)を通過させて不純物を取り除き、きれいなDNA溶液を得ます。この抽出したDNAは、特定の領域を数百万倍に増幅するPCR反応に使います。

解析する野菜リスト
これらの野菜を対象に解析をおこないます。
使用する器具
1.5 ml マイクロチューブ、遠心分離機(遠心機)、ヒートブロック(65℃)、ボルテックス、乳鉢、乳棒、乳鉢マット、液体窒素を入れるサーモカップ、薬さじ(スパテル)、マジックペン、チューブ立て、ピンセット、紫色カラム、緑色カラム、軍手
使用する試薬
DNA抽出バッファーPL1、バッファーPC、洗浄液PW1、洗浄液PW2、溶出液PE
実験1-1 :野菜からDNAを抽出する
① 野菜サンプル(以降、サンプル)の準備:葉物野菜は 3 cm× 3 cmに切り、根菜類は薄くスライスする。
② サンプルの粉砕:乳鉢に液体窒素を入れて冷やし、その中に約 100 mg のサンプルをいれ、再度液体窒素をいれて凍らせ、乳棒ですりつぶして粉末にする。



液体窒素は非常に冷たいので、利き手と反対の手に軍手をはめて乳鉢を持ちましょう。液体窒素がほぼ蒸発したタイミングが、サンプルをつぶしやすいですよ。
③ DNAの抽出:粉末状にしたサンプルを冷やした薬さじでエッペンドルフチューブに移し、400 μl のDNA抽出バッファー(PL1)を加える。その後、ボルテックスミキサーで均一になるまで(約30秒)混ぜる。

②と③のステップを手早く行うことで、より多くのDNAを抽出できます。ここはスピードを意識して進めてみましょう!
④ 酵素処理:TAの方にRNA分解酵素(RNaseA)を 10 μl 加えてもらい、再度ボルテックスミキサーで均一になるまで混ぜる。その後、ヒートブロックを65℃に設定し、10分間加温する。
⑤ 遠心分離:12,000 rpm で5分間、室温で遠心する。
⑥ 濾過:遠心後、DNAが含まれる上清をP200のマイクロピペットで紫色のカラム(Nucleo Spinfilter)に移し、12,000 rpm で2分間遠心する。その後、中のカラムを外して廃棄し、通過した液体(DNAを含む)を新しいエッペンドルフチューブに 150 μl × 2 で移す。

マイクロピペットで上清を吸い取る際には、沈殿を吸わないように注意しましょう。沈殿には不要なタンパク質や多糖類、脂質が含まれています。また、カラムを通過した液にはDNAが含まれているので、下の液体が重要です。
⑦ 洗浄:バッファーPC 450 μl を加え、ボルテックスミキサーで混ぜた後、700 μl を緑色のカラム(NucleoSpin Plant II Column)に入れ、12,000 rpmで1分間遠心する。この工程でDNAが中のカラムに付着する。その後、カラムの下にたまった液(フロースルー)はDNA以外の夾雑物(きょうざつぶつ:ゴミ)が含まれているため、廃棄する。再度カラムをチューブに差し込み、洗浄液PW1 400 μl を加えて、12,000 rpm で1分間遠心し、DNAを洗浄する。同様に洗浄液PW2 700 μl を使用して洗浄し、さらにPW2 200 μl で最終洗浄をおこなう。
⑧ DNAの溶出:溶出液PE 50 μl をカラムに加え、65℃で5分間静置した後、12,000rpmで1分間遠心する。遠心後、中のカラムを廃棄し、エッペンドルフチューブに残った溶液にDNAが含まれる。

溶出液を加えて静置することで、カラムに付着したDNAが溶け出します。遠心後、溶け出したDNAはエッペンドルフチューブに集まり、最終的に溶液として回収できます。
実験1-2:PCR反応でDNAを増幅する
次に、DNA抽出液を使用してPCR反応を行います。PCR反応では、特定のDNA領域を選び、数億〜数十億塩基対の中から数百塩基対の特異的な短い領域(バーコーディング部位)を数百万倍に増幅します。
ここでの作業はTAの方が担当し、受講者はその様子を見学しました。DNA抽出液とPCR反応液を混合したPCRチューブをサーマルサイクラー(PCR反応を起こすための機器)にセットし、反応を実施しました。

PCRで増幅させるDNAの領域は、野菜の葉緑体ゲノムの trnL-trnF 遺伝子間領域です。
▼PCRについて詳しく知りたい方はこちら
実験2:増幅したDNA断片を電気泳動で確認後、塩基配列を調べる
PCRで増幅したDNA断片のサイズを確認するために、電気泳動法を使って分離を行いました。電気泳動では、DNA分子が持つ負の電荷を利用し、電流を流すことでDNA断片を大きさごとに分けます。
まず、アガロース(寒天)ゲルを準備し、PCRで得られたDNAサンプルをゲルにあけたくぼみ(ウェル)に注入します。DNAは無色の分子ですが、特定の蛍光色素を使うことで目に見える形にすることができます。
DNAの断片は、ゲル中を移動する際、大きな断片ほど移動が遅く、小さな断片は早く移動します。これにより、異なるサイズのDNA断片を分離することができます。

電気泳動によって増幅されたDNA断片の長さを確認します。分子量マーカー(サイズを測る目安となるDNA断片)を比較に使い、結果を読み取りましょう。
▼電気泳動について詳しく知りたい方はこちら
実験に使用する器具・試薬
PCR産物、必ずバンドが見えるポジティブコントロール試料、電気泳動槽、TAEバッファー、練習用アガロースゲル、アプライ練習用サンプル、本番用アガロースゲル、マイクロピペット、チップ、撮影装置、分子量マーカー(コスモ・バイオ社 寄贈品:FastRunner DNA Ladder)
実験2-1:電気泳動
① PCRチューブに入ったPCR産物をチビタンで軽く下に落とす。
② ポジティブコントロール試料から 5 μl を吸い取り、ウェル(ゲルにあけられた穴)に注入する。
③ 調べたい他の試料(PCR産物)も同様に 5 μl をウェルに注入する。
④ 分子量マーカー 5 μl も、同じようにウェルに注入する。

⑤ 電気泳動を行い、色素がゲルの3分の2あたりに達するまで約15〜25分間泳動する。
⑥ 泳動が終了したら、撮影装置を使って結果を撮影する。
実験2-2:塩基配列の解析 – シークエンシング –
PCR反応物の残りを精製し、シークエンスプライマー(DNAの特定の領域を読み取るための開始点となる短いDNA断片)を加えた後、シークエンシングを委託業者に依頼します。業者はサンガー法を用いてDNAの正確な塩基配列を解析し、翌日にはその結果が提供されます。
具体的には、シークエンシング結果を使用して、実験3で行う相同性検索(DNAやタンパク質の配列をデータベースと照合し、類似する遺伝子や生物を探すこと)や、類似度検索(配列間の類似度を計算し、進化的な近さを数値で示すこと)を行います。これらの結果を基に、最終的に系統樹を作成し、野菜の遺伝的な関係を可視化します。
今回は時間の都合上、事前に調べておいたDNA配列データを使用して実験3(後編に掲載)に進みました。
後編に向けて
前編では、野菜からのDNA抽出、PCR反応、電気泳動を行い、DNAを増幅し、その断片の大きさを確認しました。そしてシークエンスにより各野菜の正確なDNA塩基配列を調べました。
後編では、シークエンスによって得られたDNAの塩基配列を使って、相同性検索や類似度検索を行い、野菜の遺伝的関係を調べます。さらに、これらのデータを基に系統樹を作成し、野菜同士の進化的なつながりを可視化します。
前編を通して、受講生の皆さんが真摯に実験に向き合い、試薬や器具に戸惑いながらも少しずつ使いこなせるようになった姿が印象的でした。後編でも、受講生の皆さんの更なる成長と新たな発見に期待します!



コウタイガーのちょこっと解説
電気泳動

電気泳動は、DNA分子が電流によってゲル内を移動する性質を利用して、DNA断片を大きさごとに分離する技術だよ。
DNA分子は負の電荷を持っているため、電場の中では正極に向かって移動します。アガロースゲルと呼ばれるゲル状の物質を使って電気泳動を行うと、大きなDNA断片はゲルの繊維に引っかかってゆっくりと移動し、小さなDNA断片はより速く移動します。これにより、DNAの大きさごとに異なる位置に分離されます。
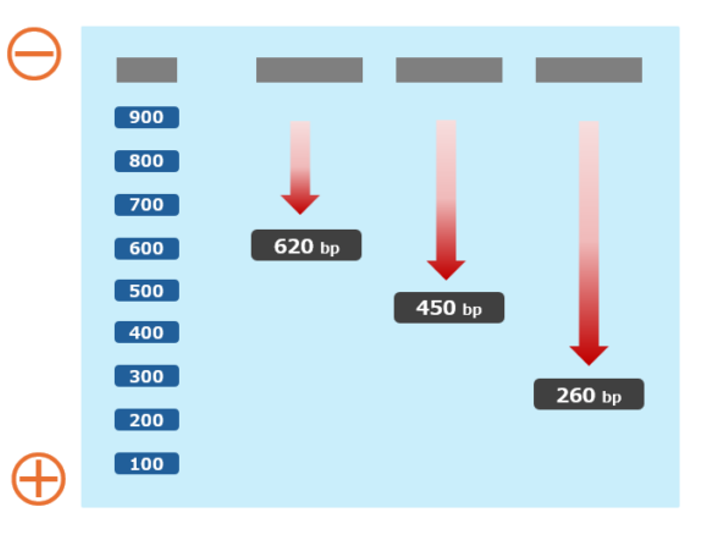
電気泳動の結果は、蛍光色素を使ってDNAを染色し、UVライトで照らすと観察することができます。DNAがゲル上にバンドとして現れるので、その位置やバンドの大きさを確認することで、PCRで増幅されたDNA断片が期待通りの長さかどうかを確認できます。
この手法は、遺伝子研究や病原体の検出などさまざまな分野で広く活用されています。
【イラスト・画像の引用元について】
本記事内で使用されたイラスト・画像は、摂南大学様のご厚意により、同大学からご共有いただいた資料の一部を引用させていただいております。

